-
[지도학습] 의사결정나무/랜덤포레스트/그래디언트 부스팅Study/Statistics 2019. 5. 24. 12:28
나무 모양으로 지도학습을 하는 모델 3가지 소개
트리의 복잡도 = 훈련데이터를 학습하기 위해 나무가 커지는 정도
의사결정나무(Decision Tree)
단계별로 예/아니오로 질문을 이어 나가면서 학습하는 모델, 스무고개마냥
리프 : 마지막 노드
순수 노드 : 0또는1로 완전히 분리 된 노드원리
종속변수를 가장 잘 나눠주는 독립변수의 특징을 가지고 데이터를 예/아니오로 분리(변수가 이항이라면 A인지B인지로, 변수가 연속형이라면 이상이하)
만약 완전히 분리 되지 않았다면 둘 중 다수에 속한 것을 예측 결과로 함
목표 : 정답에 가장 빨리 도착하는 나무 학습 => 간단하게 예측 가능하도록!
결정 트리의 복잡도 제어하기
트리 모델의 기본값은 모든 리프 노드가 순수 노드가 될 때까지 생성한 트리
=> 모든 리프 노드가 순수 노드가 될 때까지 진행하면 모델이 복잡, 훈련 데이터에 과대적합됨과대적합을 막는 방법
1. 사전가지치기 : 트리 생성을 일찍 중단하는 전략
트리 최대 깊이 or 리프의 개수 제한 or 분할하기 위한 포인트의 최소 개수 지정
2. 사후가지치기 : 트리를 만든 후 데이터 포인트(0으로 속하는지 1로 속하는지 나온 답!)가적은 노드를 삭제하거나 병합하는 전략
R에서는 지원함
의사결정 나무의 깊이 조정 이해
트리의 깊이는 특성을 몇번이나 분류할지를 정하는 것

깊이를 너무 조금 주면 거의 분류가 되지 않는 과소적합 상태가 됨

깊이를 너무 많이 주면 모델의 복잡도가 커져 시각화 하였을 때 확인도 어렵고, 훈련데이터에 과대적합 됨
매개변수
max_depht : 트리의 최대 깊이 설정
max_leaf_nodes : 리프 노드의 최대 개수 지정
max_samples_leaf : 최소 리프 노드 샘플 개수 지정
* 트리의 모델 복잡도를 조절하는 매개변수는 사전 가지치기 매개변수 중 하나만 지정해도 과대적합을 막기에 충분실습
# 기본값 설정으로 완전한 트리 생성 from sklearn.tree import DecisionTreeClassifier cancer = load_breast_cancer() X_train, X_test, y_train, y_test = train_test_split( cancer.data, cancer.target, stratify=cancer.target, random_state=42) tree = DecisionTreeClassifier(random_state=0) tree.fit(X_train, y_train) print("훈련 세트 정확도: {:.3f}".format(tree.score(X_train, y_train))) print("테스트 세트 정확도: {:.3f}".format(tree.score(X_test, y_test))) # 완전한 트리이기 때문에 훈련세트 100% 정확 > 훈련 세트 정확도: 1.000 > 테스트 세트 정확도: 0.937 # 트리의 깊이 제한 max_depth=n tree = DecisionTreeClassifier(max_depth=4, random_state=0) tree.fit(X_train, y_train) print("훈련 세트 정확도: {:.3f}".format(tree.score(X_train, y_train))) print("테스트 세트 정확도: {:.3f}".format(tree.score(X_test, y_test))) # 트리 깊이를 제한하여 훈련 세트의 정확도는 떨어졌지만 테스트 세트의 성능은 개선됨 > 훈련 세트 정확도: 0.988 > 테스트 세트 정확도: 0.951# 사전가지치기로 테스트세트 성능 개선
의사결정나무 모델링 결과를 시각화
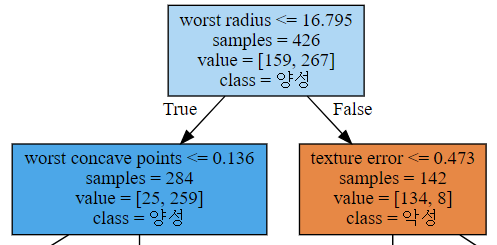
나무의 일부만 보여줌 # 'worst radius'가 16.795이하이면 왼쪽으로, 이상이면 오른쪽으로 데이터 이동
## 가장 처음에 나온 변수이며, 거의 절반에 가까운 데이터를 거르는 것을 보아 중요한 변수임을 알 수 있음
## 첫번째 노드에 포함된 samples수는 426개이며, 0으로 선택된건 159개 1로 선택된건 267개임을 알 수 있고,
1로 선택된 것이 많기 때문에 해당 노드는 양성노드로 분류됨
의사결정나무 모델의 특성 중요도를 시각화해봄
특성 중요도란?
특정 변수가 모델을 구성하는 비율을 나타냄
.feature_importances_에서 확인 가능
값이 낮다고 필요없는 변수는 아님
<= 이 트리모델이 해당 변수를 선택 하지 않아서 이거나, 다른 특성이 같은 정보를 가지고 있어서 일 수 있음
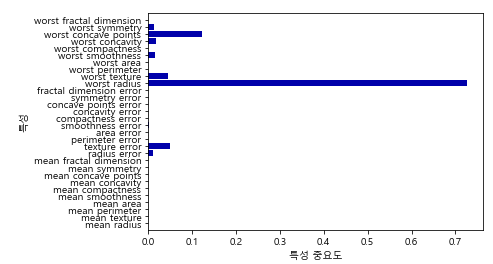
의사결정나무로 모델을 만들었을 때 특성 중요도 시각화 # 첫 노드였던 'worst radius'가 가장 중요한 특성임을 알 수 있음 => 첫 노드에서 두 클래스를 잘 나누고 있음
선형회귀와 의사결정나무의 훈련데이터, 테스트데이터 시계열 데이터 예측 정확도를 비교

트리와 선형회귀의 예측 정확도 비교 # 회귀 예측은 직선으로 훈련과 테스트 데이터 모두 꽤 정확히 예측하고 있음
# 트리 예측은 훈련데이터에 정확히 학습되어 테스트는 전혀 예측하지 못함
## 의사결정 나무는 전혀 새로운 데이터가 왔을 때 예측이 어려움을 알 수 있고
## 시계열 데이터의 경우 의사결정나무보다는 선형 회귀 예측이 적절함을 알 수 있음
장점모델을 쉽게 시각화하여 비전문가도 이해하기 쉬움(작은 트리의 경우)
데이터의 스케일에 구애받지 않음, 전처리(정규화,표준화)가 필요 없음
각 특성의 스케일이 다르거나 이진특성, 연속특성이 혼합되어 있을 때도 잘 작동
단점사전 가지치기를 사용해도 과대적합되는 경향이 있어 일반화 성능이 좋지 않음
외삽(extrapolation) = 훈련 데이터의 범위 밖의 포인트는 예측할 수 없음
어떨때 사용?
의사결정 과정을 간소하게 표현하여 비전문가에게 시각적으로 설명할 일이 있을 때
앙상블 기법
여러 머신러닝 모델을 연결하여 더 강력한 모델을 만드는 것
랜덤포레스트(Random Forest)
의사결정나무의 단점인 훈련 데이터에 과대적합을 회피할 수 있는 방법
원리
의사결정 나무의 생성을 랜덤하게 해서 Random (샘플 수 랜덤, 사용 변수 랜덤,,,)
조금씩 다른 여러 의사결정나무의 묶음이라 Forest
조금씩 다른 여러 결정 트리의 묶음?
=> 서로 다른 방향으로 과대적합된 트리를 많이 만들고 그 결과를 평균내어 과대적합된 양을 줄이기
=> 트리 모델의 예측 성능이 유지되면서 과대적합이 줄어듬모델링을 잘 하기 위해선
1. 의사결정나무를 많이 만들어야 됨
2. 각각의 트리는 타겟 예측을 잘 해야함
3. 각각의 트리는 다른 트리와 구별되어야함
트리를 랜덤하게 만드는 법
1. 트리 생성시 사용하는 데이터 포인트를 무작위로 선택하는 방법(샘플수)
2. 분할 테스트에서 특성을 무작위로 선택하는 방법랜덤포레스트 모델 구축 방법
1. 생성할 트리의 개수 정하기 (n_estimators=) 많을 수록 좋음
2. 데이터의 부트스트랩 샘플 생성
n_samples 데이터 중 무작위로 데이터를 n_samples의 횟수만큼 반복 추출어떤 데이터 포인트는 누락/중복 될 수 있음
3. 몇 개의 특성을 가지고 트리를 생성할지 정하기 (max_features=)
일반 트리처럼 전 특성 다 돌리는 게 아니고, 선택한 개수만큼만 계속 랜덤으로 돌림
값을 크게하면 : 랜덤 포레스트의 트리들은 매우 비슷해지고 의사결정나무랑 다를바가 없게됨그냥 가장 두드러진 특성이 이용됨
값을 작게하면 : 몇 개 안 선택되니까 트리들이 많이 달라지고각 트리는 데이터에 맞추기 위해 깊이가 깊어지게 됨
-> 분류하기 위해 한특성에서 계속 자르다보니까
예측방법
1. 모든 트리의 예측을 만듬
2. 회귀의 경우 : 예측들을 평균하여 최종 예측
분류의 경우 : 약한 투표 전략 사용=> 각 알고리즘이 가능성 있는 출력 레이블의 확률을 제공하여 간접적인 예측
예측한 확률을 평균내어 가장 높은 확률을 가진 클래스가 예측값이 됨
매개변수
n_estimator : 만들 트리 개수, 클수록 좋음
max_features : 사용할 특성의 수, 각 트리가 얼마나 무작위가 될 지 결정, 적게 설정하면 과대적합을 줄여줌
분류모델이면 max_features = sqrt(n_features)
회귀모델이면 max_features = n_features
추가하면 가끔 성능향상, 필요 메모리와 시간을 줄일 수 있음
n_job : 사용할 코어 수 지정 가능(=-1로 지정하면 모든 코어 사용) 모델 만들 때 시간이 오래 걸리는 경우
random_state : 지정한 값에 따라 전혀 다른 트리가 만들어져 같은 결과를 만들어야 하면 고정해야함
n_estimators로 만드는 트리 개수를 많이 지정하면 random_state값의 변화에 따른 변동이 적음
결정 트리와 마찬가지로 max_depth, max_leaf_nodes, min_sample_leaf, min_samples_split 제공실습
# two_moon데이터셋으로 트리 5개로 구성된 랜덤 포레스트 모델을 만들음 from sklearn.ensemble import RandomForestClassifier from sklearn.datasets import make_moons X, y = make_moons(n_samples=100, noise=0.25, random_state=3) X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, random_state=42) forest = RandomForestClassifier(n_estimators=5, random_state=2) forest.fit(X_train, y_train) # 랜덤 포레스트 안에 만들어진 트리는 estimator_속성에 저장 > RandomForestClassifier(bootstrap=True, class_weight=None, criterion='gini', max_depth=None, max_features='auto', max_leaf_nodes=None, min_impurity_decrease=0.0, min_impurity_split=None, min_samples_leaf=1, min_samples_split=2, min_weight_fraction_leaf=0.0, n_estimators=5, n_jobs=None, oob_score=False, random_state=2, verbose=0, warm_start=False) # 각 트리에서 학습된 결정 경계와 이를 취합해 만든 결정 경계를 함께 시각화 fig, axes = plt.subplots(2, 3, figsize=(20, 10)) for i, (ax, tree) in enumerate(zip(axes.ravel(), forest.estimators_)): ax.set_title("트리 {}".format(i)) mglearn.plots.plot_tree_partition(X, y, tree, ax=ax) mglearn.plots.plot_2d_separator(forest, X, fill=True, ax=axes[-1, -1], alpha=.4) axes[-1, -1].set_title("랜덤 포레스트") mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
시각화 결과 # 다섯개의 트리의 결정경계가 다름을 알 수 있음
# 부트스트랩 샘플링 때문에 훈련 포인트가 다르게 포함되어있어 불완전함
# 랜덤포레스트는 각 트리보다는 덜 과대적합되고 결정경계가 훨씬 좋음# 유방암 데이터셋에 100개 트리로 이뤄진 랜덤 포레스트 X_train, X_test, y_train, y_test = train_test_split( cancer.data, cancer.target, random_state=0) forest = RandomForestClassifier(n_estimators=100, random_state=0) # 100개 트리 forest.fit(X_train, y_train) print("훈련 세트 정확도: {:.3f}".format(forest.score(X_train, y_train))) print("테스트 세트 정확도: {:.3f}".format(forest.score(X_test, y_test))) # 랜덤포레스트는 아무런 매개변수 튜닝 없이도 높은 정확도를 보임 >훈련 세트 정확도: 1.000 >테스트 세트 정확도: 0.972생성한 모델의 특성 중요도를 확인해보니

랜덤 포레스트로 모델을 만들었을 때 특성 중요도 확인 # 단일 트리보다 훨씬 많은 특성이 0이상의 중요도를 가짐
# 단일 트리에서 중요했던 "worst radius"도 중요하지만 'worst perimeter'또한 중요함을 알 수 있음
장점성능이 매우 뛰어남
매개변수 튜닝 없이도 잘 작동
데이터의 스케일을 맞추지 않아도 됨 (전처리x)
매우 큰 데이터 셋에서도 잘 작동 (n_job이용하여 CPU 병렬화 가능)
단점특성의 일부만 사용해 결정 트리보다 더 깊어지는 경우가 있음
수백개의 트리를 자세히 분석하기도 어려워 => 간소하게 의사결정 과정을 표현해야하는 경우는 단일 트리
차원이 매우 높고(텍스트 데이터처럼), 희소한 데이터에서는 잘 작동하지 않음 => 이럴 경우 선형 모델 적합
많은 메모리 사용, 훈련, 예측 시 오래걸림 => 속도와 메모리에 제약이 있는 경우 선형 모델 적합
어떨때 사용?
높은 성능을 내기 때문에 대부분의 경우 사용
그래디언트 부스팅 회귀 트리(GradientBoostingRegressor)
랜덤포레스트처럼 여러 결정 트리를 묶어 강력한 모델을 만드는 앙상블 기법
이름은 회귀 트리지만 회귀, 분류 모두 사용 가능원리
얕은 트리 같은 간단한 모델(약한 학습기weak learner)을 많이 연결하는 것이 그래디언트 부스팅
-> 각 트리는 데이터의 일부만 잘 예측하기 때문에 트리가 많이 추가 될 수록 성능이 좋아짐
랜포랑 차이점
1. 이전 트리의 예측값과 타깃값 사이의 오차를 줄이는 방향으로 새로운 트리를 추가하는 알고리즘
-> 이를 위해 손실 함수를 정의하고 경사하강법을 사용하여 다음에 추가 될 트리가 예측해야 할 값을 보정
2. 무작위성이 없는 대신 강력한 사전가지치기 사용(1~5개 정도로 얕은 트리 사용) => 메모리 적게, 예측 빠르게중요 매개변수
n_estimators : 트리개수 지정=> 랜포는 클수록 좋지만, 그래디언트는 너무 커지면 모델이 복잡해지고 과대적합될 수있음
learning_rate : 이전 트리의 오차를 얼마나 강하게 보정할 것인지 제어=> 학습률이 크면 보정을 강하게 해 복잡한 모델이 만들어짐
*위의 두 트리는 깊게 연관되어 learning_rate를 낮추면 비슷한 복잡도의 모델을 만들기 위해 n_estimators를 높여야함
*가용 시간과 메모리 한도에 n_estimators를 맞추기 -> 적절한 learning_rate 찾기max_depth : 최대 트리 깊이, 각 트리의 복잡도를 낮춤, 그래디언트에서는 5이하로 설정해야함
max_leat_nodes : 리프노드의 최대개수, 각 트리의 복잡도를 낮춤실습
# 유방암 데이터 셋을 이용해 그래디언트 부스팅 실시 ## 기본값인 깊이 3, 트리수 100, 학습률 0.1 사용 from sklearn.ensemble import GradientBoostingClassifier X_train, X_test, y_train, y_test = train_test_split( cancer.data, cancer.target, random_state=0) gbrt = GradientBoostingClassifier(random_state=0) gbrt.fit(X_train, y_train) print("훈련 세트 정확도: {:.3f}".format(gbrt.score(X_train, y_train))) print("테스트 세트 정확도: {:.3f}".format(gbrt.score(X_test, y_test))) # 훈련 세트의 정확도가 100으로 과대적합 된것으로 보임 > 훈련 세트 정확도: 1.000 > 테스트 세트 정확도: 0.958과대적합을 막기 위해 트리의 최대 깊이를 줄여 사전가지치기를 강하게 하거나 학습률 낮추기
## 최대 깊이를 1로 줄이기 gbrt = GradientBoostingClassifier(random_state=0, max_depth=1) gbrt.fit(X_train, y_train) print("훈련 세트 정확도: {:.3f}".format(gbrt.score(X_train, y_train))) print("테스트 세트 정확도: {:.3f}".format(gbrt.score(X_test, y_test))) # 훈련 세트 정확도는 낮아지고, 테스트 정확도가 상승함 > 훈련 세트 정확도: 0.991 > 테스트 세트 정확도: 0.972 ## 학습률을 .01로 낮추기 gbrt = GradientBoostingClassifier(random_state=0, learning_rate=0.01) gbrt.fit(X_train, y_train) print("훈련 세트 정확도: {:.3f}".format(gbrt.score(X_train, y_train))) print("테스트 세트 정확도: {:.3f}".format(gbrt.score(X_test, y_test))) # 훈련 세트 정확도는 낮아지고, 테스트 정확도가 소폭 상승함 > 훈련 세트 정확도: 0.988 > 테스트 세트 정확도: 0.965최대 깊이를 낮추는 가지치기가 모델 성능 향상에 크게 기여함을 알 수 있음
실제로 모델링을 할 땐 가용 시간과 메모리 한도에 n_estimators를 맞추기 -> 적절한 learning_rate 찾기
생성한 모델의 특성 중요도를 확인해보니

그래디언트 부스팅으로 모델을 만들었을 때 특성 중요도 # 랜덤포레스트와 비슷하게 중요함을 확인, "worst perimeter"가 중요한 것을 알 수 있음
# 다른점은 일부 특성 중요도가 0으로 완전 무시된 것을 볼 수 있음
장점
다른 트리기반 모델처럼 특성의 스케일 조정이 필요하지 않음 (전처리x)
이진특성이나 연속적인 특성에서 잘 동작
단점매개변수를 잘 조정해야함
훈련시간이 김
트리기반 모델 특성상 희소한 고차원 데이터에는 잘 작동하지 않음어떨 때 사용할지
비슷한 종류의 데이터에서 그래디언트와 랜포 둘 다 작동하지만 더 안정적인 랜포를 먼저 사용
-> 랜포가 잘 되더라고 예측 시간이 중요하거나, 모델 성능을 쥐어짜야할 때 그래디언트 사용하면 도움됨*대규모 머신러닝에서 적용시키려면 xgboost패키지(대용량 분산 처리를 위한 오픈소스 라이브러리)와 파이썬 인터페이스를 검토하는 것이 좋음
사용한 함수 설명
트리의 특성 중요도
특성 중요도란?트리를 만드는 과정에 어떤 변수가 가장 중요한지 평가하는 것
0과1사이의 수로, 전체 합은 1, 특정 변수가 모델을 구성하는 비율을 나타냄
.feature_importances_값이 낮다고 필요없는 변수는 아님
<= 이 트리모델이 해당 변수를 선택 하지 않아서 이거나, 다른 특성이 같은 정보를 가지고 있어서 일 수 있음.feature_importances_ 에서 확인 가능
# 각 모델의 특성 중요도 시각화 (내림차순되어있지 않음) def plot_feature_importances_cancer(model): n_features = cancer.data.shape[1] plt.barh(np.arange(n_features), model.feature_importances_, align='center') plt.yticks(np.arange(n_features), cancer.feature_names) plt.xlabel("특성 중요도") plt.ylabel("특성") plt.ylim(-1, n_features) plot_feature_importances_cancer(tree)'Study > Statistics' 카테고리의 다른 글
[분석/통계] 교차검증 (0) 2019.05.29 [분석/통계] 비지도학습 - 군집(K-MEANS, 병합군집) (2) 2019.05.28 [분석/통계] 데이터 전처리와 스케일 조정 방법 (0) 2019.05.28 [지도학습] KNN (0) 2019.05.23 정규분포? (0) 2019.05.22